Die Arbeitstabelle (Matrize) wird sukzessive gelesen und durch Farb-Algorithmus 1 in eine Tabelle von äquivalenten Wellenlängen und RGB-Werten umgewandelt.
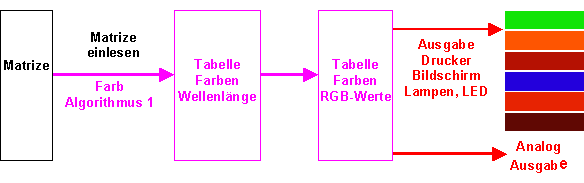
Bestimmung der Farben für die Basentripletts
Legt man die liniearisierte Tabelle der Tripletts neben das Lichtspektrum, so lassen sich die Tripletts direkt in Farben linear übertragen. Legt man das Triplett aaa an das rote Ende und das Triplett ttt an das blaue Ende, dann lassen sich die Wellenlängen L für die zugeordneten Farben aus dem Positionswert eines Tripletts errechnen.
Bestimmung der Spektralwertfunktionen
Für die Darstellung der Farben über einen Bildschirm, Drucker oder anderen optischen Ausgabegeräten wie LEDs müssen noch die RGB-Werte für die ermittelten Farben berechnet werden.Dabei sind die sogenannten Spektralwertkurven r(L), g(L), b(L) von Bedeutung, die die Intensität (Tristimulus) der drei Grundfarben (rot, grün, blau) in Bezug zur Wellenlänge wiedergeben.
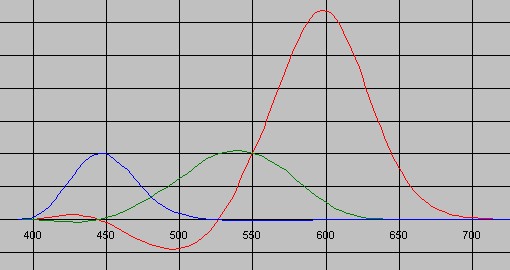
Für die Spektralwertkurven gibt es keine mathematischen Gleichungen, dafür aber Tabellen. Aus einer Tabelle der Spektralwertkurven für die obige Abbildung lässt sich, durch eine numerische Fourieranalyse, eine Gleichung für jede der drei Funktionen r(L), g(L), b(L) gewinnen.
Bestimmung der RGB-Werte
Die einfachste Art der Umwandlung der Spektralwertkurven in eine RGB-Farbpalette besteht zuerst darin die Spektralwertfunktionen bzgl. ihrer Werte zu normieren. Der Farb-Algorithmus 1 liefert folgendes Farbspektrum:| aaa | caa | gaa | taa | ||||
| aac | cac | gac | tac | ||||
| aag | cag | gag | tag | ||||
| aat | cat | gat | tat | ||||
| aca | cca | gca | tca | ||||
| acc | ccc | gcc | tcc | ||||
| acg | ccg | gcg | tcg | ||||
| act | cct | gct | tct | ||||
| aga | cga | gga | tga | ||||
| agc | cgc | ggc | tgc | ||||
| agg | cgg | ggg | tgg | ||||
| agt | cgt | ggt | tgt | ||||
| ata | cta | gta | tta | ||||
| atc | ctc | gtc | ttc | ||||
| atg | ctg | gtg | ttg | ||||
| att | ctt | gtt | ttt |
Zweistrangbehandlung
Wird nicht nur ein Basenstrang sondern der komplette DNA-Strang verarbeitet, dann benötigt man die Komplementärtripletts.Durch die Linearisierung der DNA-Tripletts ist der Positionswert des Komplementärtripletts bereits bekannt. Durch die vorhandenen Farbgleichungen lassen sich daher die Farbwerte bzw. Wellenlängen des Tripletts sowie des Komplementärtripletts sofort ermitteln.
Da der RGB-Wert für das zugrunde liegende Triplett vorhanden ist, lässt sich bei hexadezimaler Handhabung der RGB-Farbe (z.B. Computeranwendung oder einem Bildschirm) eine einfache Konvertierung ermöglichen. Es braucht nur das hexadezimale (binäre) Komplement der RGB-Farbe gebildet werden, um die Komplementfarbe zu erzeugen.
Anwendung Farb-Algorithmus 1
Im Farb-Algorithmus 1 werden den Basen a, c, g ,t sichtbare Farben des elektromagnetischen Frequenzspektrums zugeordnet, die dann in einen spektralen RGB-Farbraum übersetzt werden.Die folgende Abbildung zeigt den Anfang (die ersten 26 Tripletts) aus dem kompletten mitochondrialen Genom eines Neandertalers (Homo sapiens neanderthalensis) auf Grundlage von Farb-Algorithmus 1.

Durch den Farb-Algorithmus 1 (Spektrale Farbzuordnung) ist gewährleistet, dass eine lineare (sogar isomorphe, also eineindeutige) Abbildung zwischen DNA und Farben vorhanden ist. Die erzeugten Farben können daher als reale Entsprechung der DNA auf einer optischen Wahrnehmungsebene aufgefasst werden.
